使用用 SVM 进行乳腺癌检测
在了解了如何创建和使用 SVM 分类器后,我们来看一个实际的项目,数据集来自美国威斯康星州的乳腺癌诊断数据集,点击这里进行下载。
医疗人员采集了患者乳腺肿块经过细针穿刺 (FNA) 后的数字化图像,并且对这些数字图像进行了特征提取,这些特征可以描述图像中的细胞核呈现。肿瘤可以分成良性和恶性。 数据表一共包括了 32 个字段,代表的含义如下:
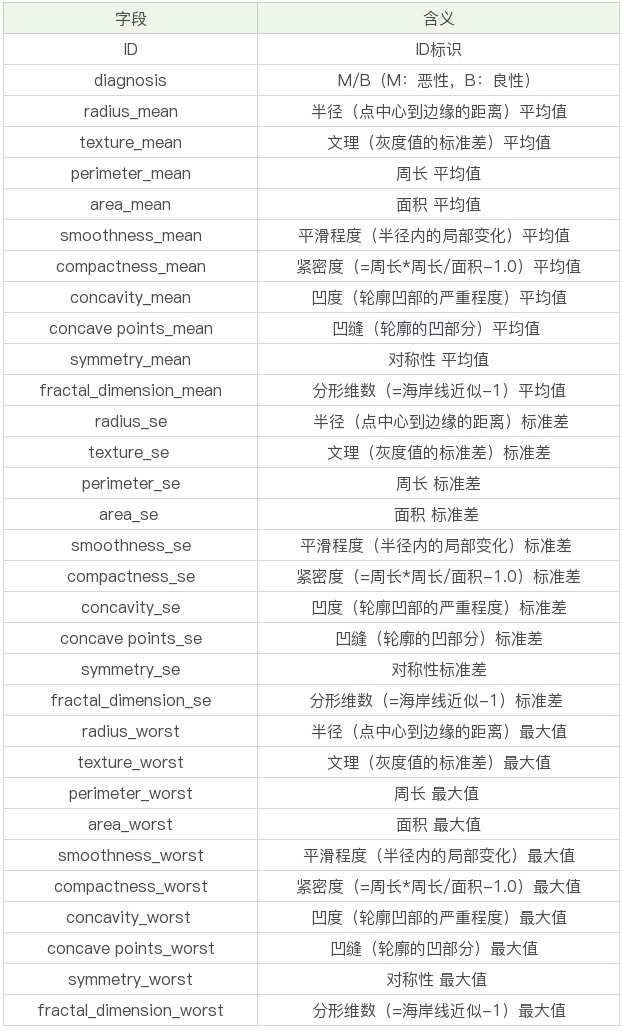
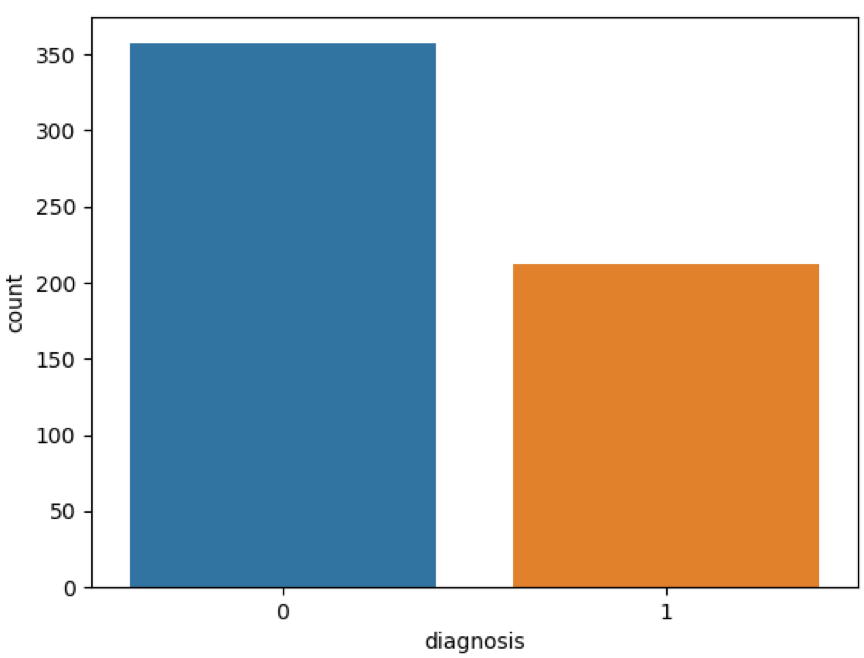
上面的表格中,mean 代表平均值,se 代表标准差,worst 代表最大值(3 个最大值的平均值)。每张图像都计算了相应的特征,得出了这 30 个特征值(不包括 ID 字段和分类标识结果字段 diagnosis),实际上是 10 个特征值(radius、texture、perimeter、area、smoothness、compactness、concavity、concave points、symmetry 和 fractal_dimension_mean)的 3 个维度,平均、标准差和最大值。这些特征值都保留了 4 位数字。 字段中没有缺失的值。在 569 个患者中,一共有 357 个是良性,212 个是恶性。
好了,我们的目标是生成一个乳腺癌诊断的 SVM 分类器,并计算这个分类器的准确率。首先设定项目的执行流程:
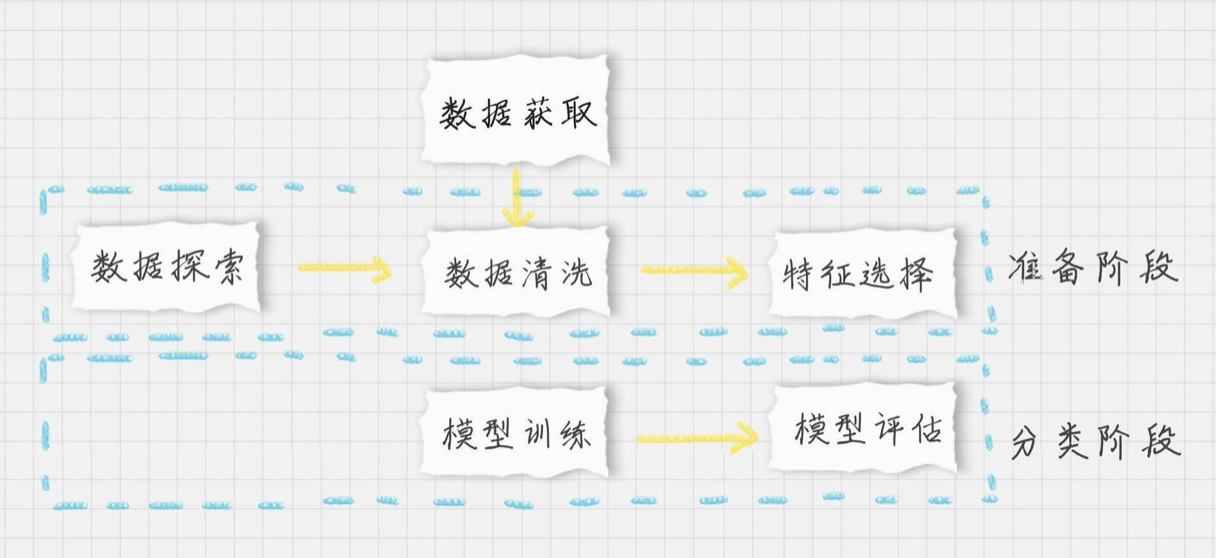
运行步骤
- 1.获取数据
- 2.数据基本处理
- 2.1 确定特征值,目标值
- 2.2 缺失值处理
- 2.3 数据集划分
- 3.特征工程(字典特征抽取)
- 4.机器学习(决策树)
- 5.模型评估- 首先我们需要加载数据源;
- 在准备阶段,需要对加载的数据源进行探索,查看样本特征和特征值,这个过程你也可以使用数据可视化, 它可以方便我们对数据及数据之间的关系进一步加深了解。然后按照“完全合一”的准则来评估数据的质量,如果数据质量不高就需要做数据清洗。 数据清洗之后,你可以做特征选择,方便后续的模型训练;
- 在分类阶段,选择核函数进行训练,如果不知道数据是否为线性,可以考虑使用 SVC(kernel=‘rbf’) ,也就是高斯核函数的 SVM 分类器。然后对训练好的模型用测试集进行评估。
按照上面的流程,我们来编写下代码,加载数据并对数据做部分的探索:
# 乳腺癌诊断分类
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split
from sklearn import svm
from sklearn import metrics
from sklearn.preprocessing import StandardScaler
# 加载数据集,你需要把数据放到目录中
data = pd.read_csv("data.csv")
# 因为数据集中列比较多,我们需要把 dataframe 中的列全部显示出来
pd.set_option('display.max_columns', 1000)
pd.set_option('display.width', 1000)
pd.set_option('display.max_colwidth', 1000)
# 数据探索
print(data.columns)
print(data.head(5))
print(data.describe())这是部分的运行结果,完整结果你可以自己跑一下。
Index(['id', 'diagnosis', 'radius_mean', 'texture_mean', 'perimeter_mean',
'area_mean', 'smoothness_mean', 'compactness_mean', 'concavity_mean',
'concave points_mean', 'symmetry_mean', 'fractal_dimension_mean',
'radius_se', 'texture_se', 'perimeter_se', 'area_se', 'smoothness_se',
'compactness_se', 'concavity_se', 'concave points_se', 'symmetry_se',
'fractal_dimension_se', 'radius_worst', 'texture_worst',
'perimeter_worst', 'area_worst', 'smoothness_worst',
'compactness_worst', 'concavity_worst', 'concave points_worst',
'symmetry_worst', 'fractal_dimension_worst'],
dtype='object')
id diagnosis radius_mean texture_mean perimeter_mean area_mean \
0 842302 M 17.99 10.38 122.80 1001.0
1 842517 M 20.57 17.77 132.90 1326.0
2 84300903 M 19.69 21.25 130.00 1203.0
3 84348301 M 11.42 20.38 77.58 386.1
4 84358402 M 20.29 14.34 135.10 1297.0接下来,我们就要对数据进行清洗了。
运行结果中,你能看到 32 个字段里,id 是没有实际含义的,可以去掉。diagnosis 字段的取值为 B 或者 M,我们可以用 0 和 1 来替代。另外其余的 30 个字段,其实可以分成三组字段,下划线后面的 mean、se 和 worst 代表了每组字段不同的度量方式,分别是平均值、标准差和最大值。
# 将特征字段分成 3 组
features_mean = list(data.columns[2:12])
features_se = list(data.columns[12:22])
features_worst = list(data.columns[22:32])
# 数据清洗
# ID 列没有用,删除该列
data.drop("id", axis=1, inplace=True)
# 将 B 良性替换为 0,M 恶性替换为 1
data['diagnosis'] = data['diagnosis'].map({'M': 1, 'B': 0})然后我们要做特征字段的筛选,首先需要观察下 features_mean 各变量之间的关系,这里我们可以用 DataFrame 的 corr() 函数,然后用热力图帮我们可视化呈现。同样,我们也会看整体良性、恶性肿瘤的诊断情况。
# 将肿瘤诊断结果可视化
sns.countplot(data['diagnosis'], label="Count")
plt.show()
# 用热力图呈现 features_mean 字段之间的相关性
corr = data[features_mean].corr()
plt.figure(figsize=(14, 14))
# annot=True 显示每个方格的数据
sns.heatmap(corr, annot=True)
plt.show()这是运行的结果:
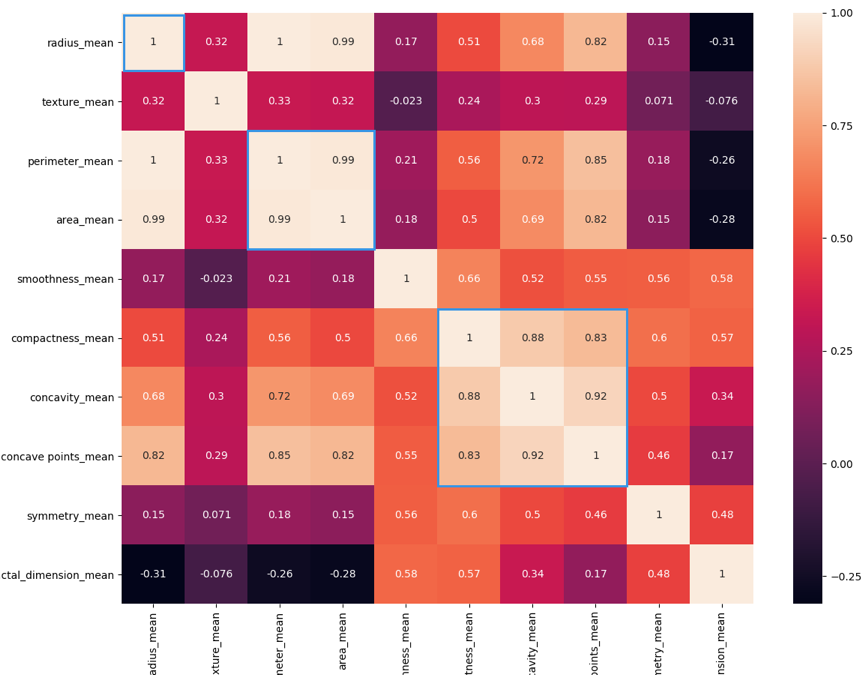
热力图中对角线上的为单变量自身的相关系数是 1。颜色越浅代表相关性越大。所以你能看出来 radius_mean、perimeter_mean 和 area_mean 相关性非常大,compactness_mean、concavity_mean、concave_points_mean 这三个字段也是相关的,因此我们可以取其中的一个作为代表。
那么如何进行特征选择呢?
特征选择的目的是降维,用少量的特征代表数据的特性,这样也可以增强分类器的泛化能力,避免数据过拟合。
我们能看到 mean、se 和 worst 这三组特征是对同一组内容的不同度量方式,我们可以保留 mean 这组特征,在特征选择中忽略掉 se 和 worst。同时我们能看到 mean 这组特征中,radius_mean、perimeter_mean、area_mean 这三个属性相关性大,compactness_mean、daconcavity_mean、concave points_mean 这三个属性相关性大。我们分别从这 2 类中选择 1 个属性作为代表,比如 radius_mean 和 compactness_mean。
这样我们就可以把原来的 10 个属性缩减为 6 个属性,代码如下:
# 抽取特征选择的数值作为训练和测试数据
X = data[['radius_mean', 'texture_mean',
'smoothness_mean', 'compactness_mean',
'symmetry_mean', 'fractal_dimension_mean']]
y = data['diagnosis']对特征进行选择之后,我们就可以准备训练集和测试集:
# 抽取 30% 的数据作为测试集,其余作为训练集
x_train, x_test, y_train, y_test = train_test_split(X, y, test_size=0.3)在训练之前,我们需要对数据进行规范化,这样让数据同在同一个量级上,避免因为维度问题造成数据误差:
# 采用 Z-Score 规范化数据,保证每个特征维度的数据均值为 0,方差为 1
ss = StandardScaler()
train_X = ss.fit_transform(x_train)
test_X = ss.transform(x_test)最后我们可以让 SVM 做训练和预测了:
# 创建 SVM 分类器
# model = svm.SVC()
model = svm.LinearSVC()
# 用训练集做训练
model.fit(train_X, y_train)
# 用测试集做预测
prediction = model.predict(test_X)
print('准确率:', metrics.accuracy_score(prediction, y_test))运行结果:
准确率: 0.9181286549707602准确率大于 90%,说明训练结果还不错。